Total solutions for every patient
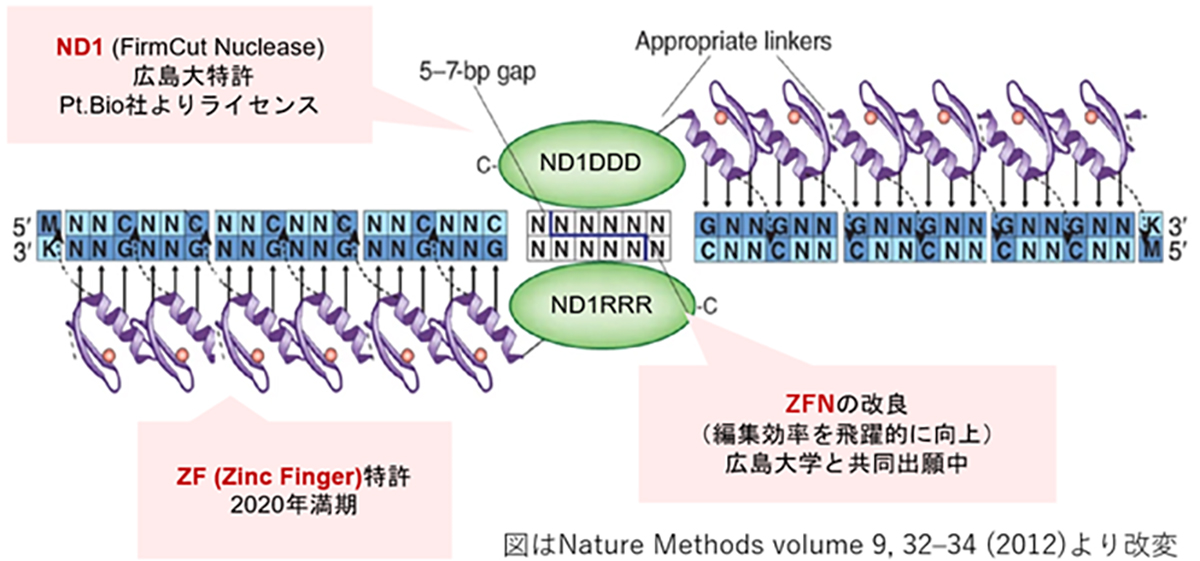
Introduced in 1996, ZFN is the first-generation genome editing enzyme, an artificial protein blending the DNA-binding domain called the Zinc finger motif with the nuclease domain. A single Zinc finger can recognize three base pairs of DNA. By linking multiple Zinc fingers, you can produce a Zinc finger that recognizes extended DNA sequences and binds specifically to target sequences. As endonucleases operate as dimers, when two ZFNs bind to their respective target sequences, a nuclease dimer forms, resulting in DNA cleavage.
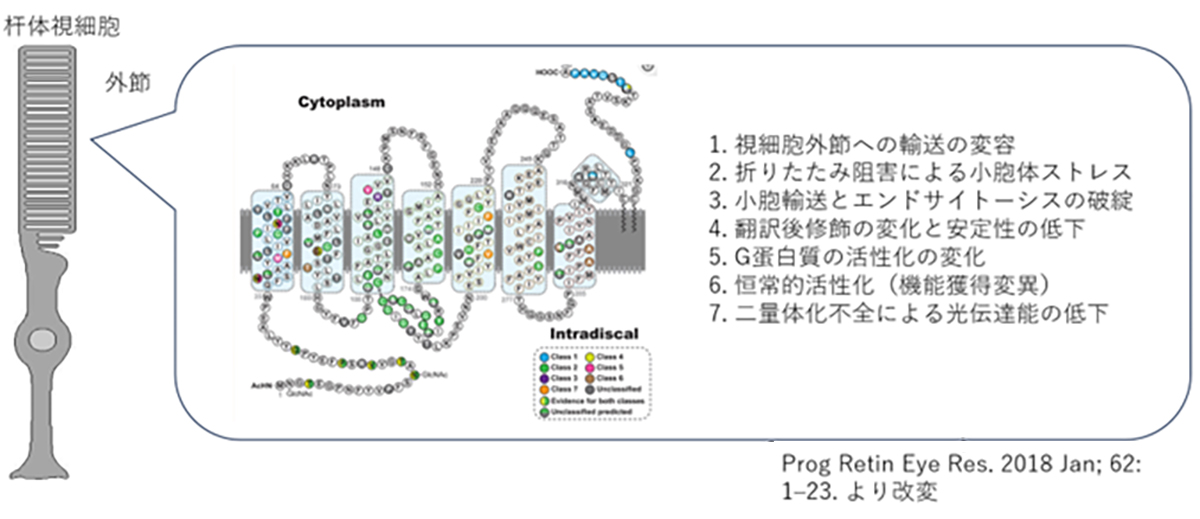
Rhodopsin (RHO) is a light-sensing protein situated in a part of the rod photoreceptor cells called the outer segment. This gene has evolved to detect even minimal light, and rhodopsin constitutes about 90% of the total protein in the outer segment. The capability of rod cells to perceive light in dim or nighttime environments is heavily reliant on rhodopsin’s functionality. Rhodopsin (RHO) gene mutations account for approximately 30% of the dominant forms of Retinitis Pigmentosa, with an estimated 100,000 patients worldwide.
Retinitis Pigmentosa (RP) is a set of genetic disorders leading to the gradual degeneration of rod cells, the photoreceptors responsible for vision in dim light. As the condition worsens, it can start to affect cone cells, which manage central vision, color perception, and sharpness of vision, possibly culminating in complete blindness. Currently, there are more than 250 identified genes associated with RP, although the specific function of many of these genes is not yet fully understood. The pattern of inheritance can vary greatly depending on the causative gene, with all forms being reported: autosomal dominant, autosomal recessive, and X-linked. However, there are also isolated cases where no other instances of the condition can be traced within the patient’s family.
The onset of Retinitis Pigmentosa can vary based on the causative gene and environmental factors, but it typically manifests between the ages of 20 to 40, and symptoms gradually progress over a span of 5 to 10 years (though some individuals might exhibit symptoms as early as childhood or in their teens).
ロドプシン(RHO)は杆体視細胞の外節と呼ばれる部分に局在する光受容タンパク質で、僅かな光でも感知できるように遺伝子が進化しており、また外節部分の総タンパク質の9割をロドプシンが占めています。杆体視細胞が夜間や暗いところで光を感知する特徴はロドプシンの機能に大きく依存しています。ロドプシン(RHO)遺伝子変異は顕性型の網膜色素変性の原因遺伝子の約3割を占め、世界では10万人程度の患者さんが推定されています。
ZFNは1996年に発表された第1世代のゲノム編集因子で、Zinc fingerモチーフと呼ばれるDNA結合ドメインにヌクレアーゼドメインを結合した人工タンパク質です。1つのZinc fingerは3塩基対のDNAを認識するので、Zinc fingerを複数連結すると長いDNA配列を認識し標的配列に特異的に結合するZinc fingerを作製できます。ヌクレアーゼドメインは2量体で機能するエンドヌクレアーゼが用いるため、2つのZFNを作製して各標的配列に結合した時にヌクレアーゼ2量体が形成される事によりDNAが切断されます。
網膜色素変性(RP)は、遺伝的背景により暗所での視機能を司る桿体視細胞が進行性に変性する疾患群であり、さらに進行すると中心視野、視力、色覚など司る錐体視細胞も2次的な変性をきたし、完全失明に至ることもある疾患です。現在では250もの原因遺伝子が報告されていますが、それらの遺伝子機能が解っていないものが多いです。その遺伝形式については常染色体優性、常染色体劣性、X連鎖性など、原因遺伝子により全ての形式が報告されていますが、家系内に他の発症者が確認できない孤発例もあります。原因遺伝子や生活環境によりますが、おおよそ20代〜40代で発症し、5年から10年に掛けて徐々に症状が進行します(幼少期から10代で発症される方もおられます)。
Introduced in 1996, ZFN is the first-generation genome editing enzyme, an artificial protein blending the DNA-binding domain called the Zinc finger motif with the nuclease domain. A single Zinc finger can recognize three base pairs of DNA. By linking multiple Zinc fingers, you can produce a Zinc finger that recognizes extended DNA sequences and binds specifically to target sequences. As endonucleases operate as dimers, when two ZFNs bind to their respective target sequences, a nuclease dimer forms, resulting in DNA cleavage.
While ZFNs offer the advantage of a smaller molecular weight, making them easier to load onto vectors like AAV, designing a highly specific ZFN for a target sequence is challenging and time-consuming. However, our joint research with Hiroshima University has achieved high-throughput development of highly specific ZFNs. Leveraging Hiroshima University’s FirmCut nuclease ND1, we established ZFN development technology that achieves a cleavage efficiency comparable to CRISPR-Cas9 in adult retinal cells.
In developing therapeutic agents through HITI gene insertion, it’s vital to craft the right constructs to ensure peak gene functionality, pinpoint the optimal vector for gene delivery, and hone the insertion techniques. Drawing from insights in functional genomics and stem cell development, our research diligently focuses on designing constructs for optimized gene functionality. Collaboratively working with Simprogen Corp. in Kobe, we’re pioneering the development of high-quality adenoviral vectors. Moreover, in partnership with the Kobe Eye Center Hospital, we’re advancing our understanding and methodology of precise gene insertion techniques.
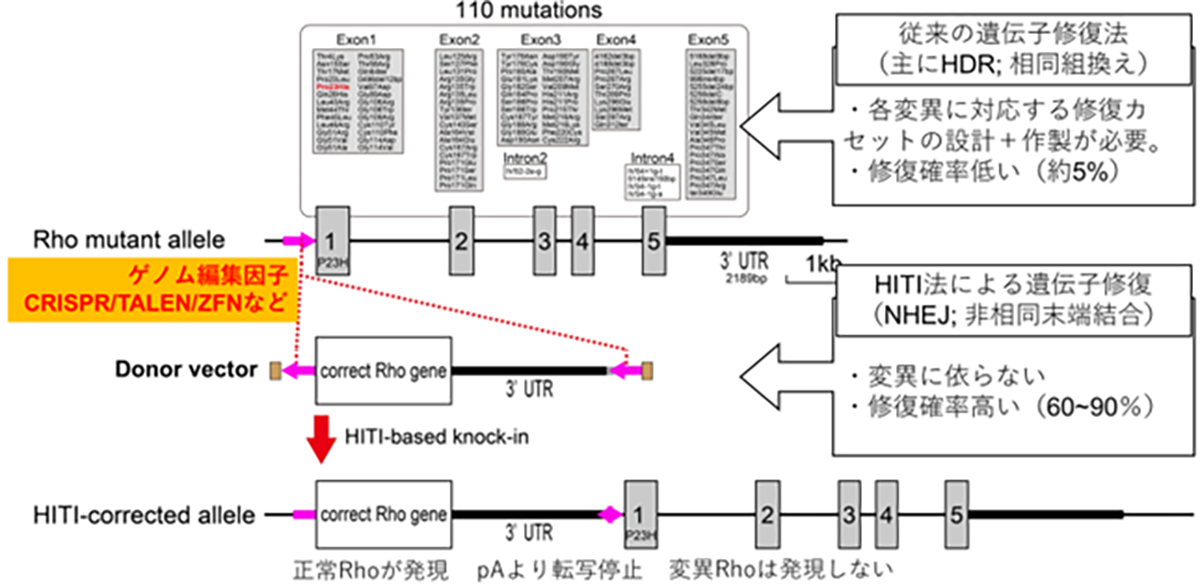
For dominant mutations, such as those targeting rhodopsin, a gene therapy remedy needs to address both the repair of the causative mutation causing dominant inhibition and the replenishment of normal RHO gene expression. Since there are 110 potential mutation sites in the rhodopsin gene locus, it’s more efficient to insert (or replace with) the full-length normal rhodopsin gene rather than develop a separate gene therapy for each mutation. This way, one treatment can potentially address all mutations, including any new ones.
When introducing external, normal-sequence DNA, the common approach utilizes HDR-based genome editing. A challenge arises since HDR is prevalent in dividing cells but less so in non-dividing cells, and the majority of adult tissue cells are non-dividing. To address this, the HITI (Homology-independent targeted integration) method was developed. It leverages the NHEJ repair pathway, notable for its high efficiency in non-dividing cells, to accurately insert foreign genes. This makes gene insertion in specific sequences of adult cells, such as retinal cells, achievable. By positioning the normal sequence DNA just prior to the start codon of a mutated gene locus, the normal gene is expressed using the original gene’s promoter, suppressing the mutated gene’s expression. Essentially, this method simultaneously rectifies dominant mutations causing dominant inhibition and restores the expression of the normal RHO gene.
Genome editing, which allows rewriting of cellular genetic information, holds great promise for treating gene mutations that manifest due to dominant inhibition. This technique targets specific sequences in DNA or its transcription product, mRNA, to introduce changes.
The enzymes employed in genome editing come in three main categories or generations:
The commonly utilized genome editing approach involves “cutting” at the target sequence. The induced cuts exploit two intrinsic cellular gene repair mechanisms to insert (recombine) genes or introduce mutations.
ZFNは1996年に発表された第1世代のゲノム編集因子で、Zinc fingerモチーフと呼ばれるDNA結合ドメインにヌクレアーゼドメインを結合した人工タンパク質です。1つのZinc fingerは3塩基対のDNAを認識するので、Zinc fingerを複数連結すると長いDNA配列を認識し標的配列に特異的に結合するZinc fingerを作製できます。ヌクレアーゼドメインは2量体で機能するエンドヌクレアーゼが用いるため、図のように2つのZFNを作製して各標的配列に結合した時にヌクレアーゼ2量体が形成される事によりDNAが切断されます。
ZFNは、CRISPRやTALENよりも分子量が小さくAAV等のベクター(運搬体)に載せやすい利点がある反面、目的配列に特異性の高いZFNの設計が難しく作製に時間を要するため、開発の大きな律速となっていました。我々は、広島大学との共同研究より特異性の高いZFN開発のハイスループット化を実現し、また同大学が開発したFirmCutヌクレアーゼND1を用いる事等により、成体視細胞でCRISPR-Cas9と同等の切断効率を持つZFNの開発技術を確立しました。
HITI遺伝子挿入による治療製剤の開発には、遺伝子が効率的に機能するためのコンストラクト設計、遺伝子を送達するベクター(運搬体)、そして導入手法の最適化が重要です。我々の研究では、機能ゲノム学と幹細胞発生学の知見を基にして、遺伝子が効率的に機能するためのコンストラクトを設計しています。また、シンプロジェン社(神戸市)と協力し、高品質のアデノ随伴ウイルスベクターの開発に取り組むとともに、神戸アイセンター病院と連携し適切な導入手法の確立のための研究を進めています。

図 ZFNの遺伝子認識イメージと、実験による編集効率の向上
顕性型変異、例えばロドプシンを標的とした原因遺伝子治療製剤には、優性阻害となる原因変異の修復と、正常RHO遺伝子発現の補充の両方への対処が必要となります。ロドプシン遺伝子座には110カ所の変異があるため、各変異に対する遺伝子治療を個別に開発するよりも正常な全長のロドプシン遺伝子を挿入(置換)する方法がより効率的で、1種類の製剤で新規の変異を含む全ての変異を治療する事が可能となります。
正常配列DNAを外から供給する場合はHDRを介したゲノム編集技術が一般的ですが、HDRは分裂細胞での頻度が高く非分裂細胞では低い傾向があります。成体の組織を構成するほとんどの細胞は非分裂細胞であることから、成体細胞に対してのDNAを導入・置換の効率が低い事が課題でした。HITI(Homology-independent targeted integration)法による遺伝子挿入法は、非分裂細胞でも頻度が高いNHEJ修復経路を利用して外来の遺伝子を正しい方向で挿入する技術であり、網膜細胞を含む成体細胞でも高頻度で目的配列に遺伝子を挿入する事が可能となります。変異を持つ遺伝子座の翻訳開始部分(開始コドン)前に正常配列DNA挿入する事で、遺伝子本来のプロモータから挿入した正常遺伝子を発現させつつ、変異遺伝子の発現が停止します。すなわち、顕性型変異における優性阻害となる原因遺伝子の修復と正常RHO遺伝子発現の補充の両方の対処が可能となります。

HITI遺伝子導入の図解
顕性遺伝型のうち特に優性阻害により発症する遺伝子変異に対しては、ゲノム編集技術による治療法が高い効果を期待されています。ゲノム編集は細胞の遺伝情報(ゲノム)を書き換える技術の総称であり、ゲノム本体であるDNAや転写産物のmRNAの特定の配列を狙って変化させることが可能となります。
ゲノム編集を行う酵素はゲノム編集因子と呼ばれ、標的配列をデザインできるゲノム編集因子は第1世代のZFN(Zinc finger nuclease)、第2世代のTALEN(Transcription activator-like effector nuclease)、第3世代のCRISPR(Clustered regularly interspaced short palindromic repeats)に大別されます。ZFNは様々な転写因子のDNA結合モチーフが基となっており、1つのモチーフで3塩基対のDNAを認識します。TALENは細菌由来のDNA結合モチーフが基となっており、1つのモチーフで1つのDNAを認識するため、ZFNより配列が長くなりますが特異性の高いゲノム編集因子の設計が可能となりました。CRISPRは細菌の免疫機構(CRISPR/Casシステム)が基になっており、Casヌクレアーゼと標的配列を認識するsgRNA(1本鎖ガイドRNA)で構成されます。タンパク質モチーフで塩基配列を認識するZFN, TALENに比べ、CRISPRは標的配列の認識をRNA-DNA相互作用によって行うため、標的配列の予測や構築が容易になりゲノム編集の研究応用が飛躍的に進みました。
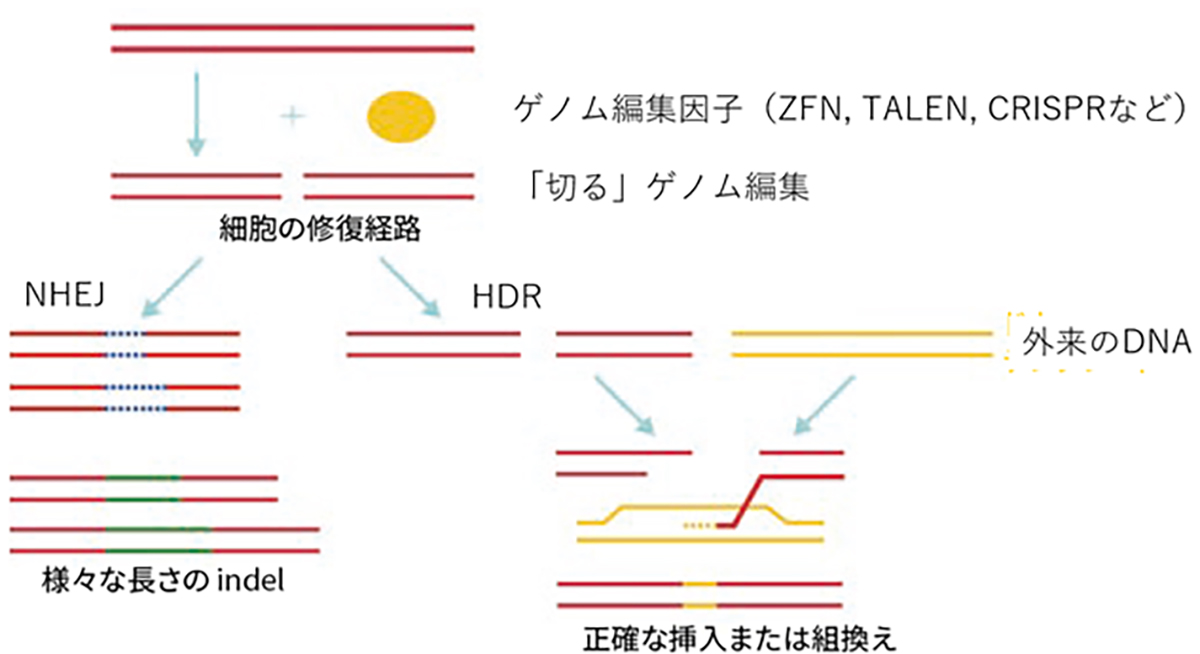
現在頻用されるゲノム編集は狙った配列を「切る」編集で、切断に伴い誘導される細胞内の2つの遺伝子修復機構を利用して、遺伝子を挿入(組み換える)編集や変異を加える編集を行います。遺伝子を挿入する編集は、相同組換え修復(homology-directed repair; HDR)と呼ばれる対立遺伝子から修復遺伝子を合成し組換える修復を利用し、切断時に正常配列を持つ鋳型DNAを外来から供給し組み替える事で正確なゲノム編集が期待できます。変異を加える編集は、非相同末端結合(non-homologous end-joining; NHEJ)と呼ばれる、切断されたDNAの両端を任意の塩基の挿入・欠失(InDel)を伴いながら結合する修復機構を用います。InDelは任意であるため標的遺伝子の改変(主にノックアウト)が起こります。

図:ゲノム編集因子と、「切る」ゲノム編集からできる事(HDRとNHEJ)
ロドプシン(RHO)は杆体視細胞の外節と呼ばれる部分に局在する光受容タンパク質で、僅かな光でも感知できるように遺伝子が進化しており、また外節部分の総タンパク質の9割をロドプシンが占めています。杆体視細胞が夜間や暗いところで光を感知する特徴はロドプシンの機能に大きく依存しています。ロドプシン(RHO)遺伝子変異は顕性型の網膜色素変性の原因遺伝子の約3割を占め、世界では10万人程度の患者さんが推定されています。
ヒトのロドプシンは348のアミノ酸から構成されていますが、これまでの遺伝子診断より110カ所の異なる変異が報告されています。変異遺伝子のほとんどが優性阻害/機能獲得変異を示しますが、機能変容は変異場所による事が知られており、光感受性の低下や恒常的活性化やロドプシンタンパク質の輸送不全などがあります。また阻害効果も変異により異なるため、低分子化合物などを用いての対症療法には適切な薬剤処方が必要です。加えてロドプシン遺伝子はハプロ不全でも発症する原因遺伝子であるため、RHOを標的とした原因遺伝子治療製剤には優性阻害となる原因変異の修復と、正常RHO遺伝子発現の補充の両方への対処が必要です。